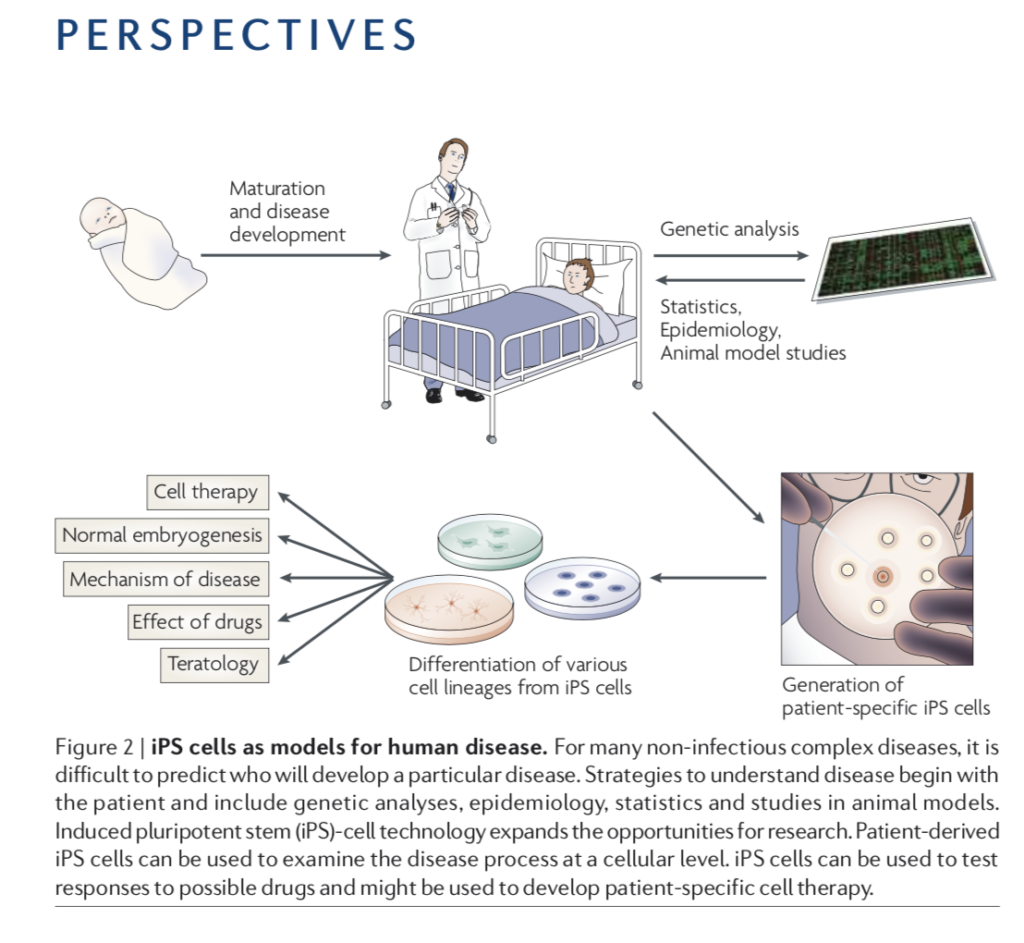
図は、山中さんがヒトiPS作成に成功したあと、私がNature Review Molecular Cell Biologyに米国の1型糖尿病患者団体JDRFの友人と一緒に描いた、iPSへの期待についての論文から転載した図だが、この時患者さんからiPSを作成し、一方でゲノム解析を行って、疾患の成立メカニズムを細胞レベルで調べる研究が大きく発展するのではないかと期待した。この時CRISPR技術は念頭になかったが、その後この技術で遺伝子変異を再構成して調べることも可能になっている。
今日紹介するニューヨーク・マウントサイナイ病院からの論文は、正常iPSに統合失調症と強く相関が見られるSNPや発現量の違いを再構成しようとする研究で疾患iPSを超える可能性にチャレンジした論文だが、読んだ後やはり病気の再構成は統合失調症のような多くの遺伝子が関わる病気については、先は長いという印象を持った。タイトルは「Synergistic effects of common schizophrenia risk variants(統合失調症リスクのコモンバリアント変異は協調的効果を持つ)」だ。
現在まで統合失調症と相関が認められる遺伝子変異は150種類に達しようとしている。異なる遺伝子でも、小さな変異が集まって共通の病気を誘導していると思われるが、さすがにこの小さな変異を集めてという点を細胞レベルで再現するのは難しい。そこで、この研究では、コモンバリアントの中でも特に遺伝子発現の差としてはっきり相関性が認められる遺伝子をまず探し、様々な前駆体タンパク質を活性化するセリンプロテアーゼFurin、およびシナプス形成に関わるSNAP91、TSNARE1に焦点を合わせることを決めている。ただ、この選択は恣意性がないとは言えないと思う。
さて、一番正しい再構成は統合失調症と相関するSNPをゲノムに導入することで、著者らもこれに挑戦しているが、これはまだまだ簡単ではない。というのも、今のところSNPを導入した細胞を選択するのは、個々の細胞の遺伝子を調べる以外に方法はなく、いくらCRISPRでも大変な実験なようだ。最終的には苦労が認められ、このサイトでAAがGGになると、細胞や分化時期で発現が大きく変化することが示され、また神経突起の伸長に異常があることを明らかにしている。すなわち、iPS を用いるとコモンバリアントの細胞レベルでの効果を確かに調べられる。
残念ながら、SNPを導入したのはfurinだけで、あとは細胞レベルの発現の違いをCRISPRで抑制したり、活性化したりする方法で、シナプス形成分子の発現を変えることで、たしかにシナプスでの興奮性が変化することが確認できる。ただ、furinのデータと比べると、インパクトは弱い。
そこで最後に、furinのSNPと、他3種類のシナプス形成分子の発現異常を組み合わせて導入した細胞を、それぞれの変異を持った細胞と比べ、統合失調症の変化を再構成されるか調べている。また、統合失調症患者をはじめとする様々な病気の患者さんの脳細胞のデータと遺伝子発現について比べている。
この再構成実験では機能的検討ができておらず、遺伝子発現のパターンで再現どを評価しているが、全てを合わせたことで、様々な疾患とより強い正の相関を得られることを示している。
結局多くのデータをうまくまとめたという印象があり、またSNP自体を導入するのはやはり大変な実験なので、遺伝子変異を細胞内に再構成するという道のりはまだまだだと思った。その意味で、2008年に期待しように、多くの遺伝子型の細胞を集めて分析することも意外と近道だと確信した。



病気の再構成は統合失調症のような多くの遺伝子が関わる病気については、先は長いという印象を
Imp:
iPS細胞の興味深い応用例だと思います。病気の異常遺伝子をiPS細胞内で再構成し研究する。