核酸配列を使ったバーコードを用いて、Single cell レベルでトランスクリプトームやクロマチンの状態を調べる方法は、生命科学分野の大きな変革をもたらしたと思う。このバーコード技術を、組織学と合体する試みも進んでおり、最初に紹介したのは今から5年以上前の2016年で、バーコード付きの RNA トラップを、スライドグラスにタイル状に張り巡らせ、バーコードから二次元的位置関係を再構成して、組織情報と統合するという方法で(https://aasj.jp/news/watch/5490)、その後同じような方法を用いた研究も何回か紹介した。
ただ、single cell を液滴にトラップする方法と異なり、このような二次元スポットを用いる方法では、標的の核酸を調整するためにプロセスする方法は使えない。そこで考え出されたのが、組織切片上で様々な処理をした後、バーコードを後から結合させる方法で、最初はX軸、その後Y軸と異なるバーコードを組織上で反応させて、処理された標的核酸をバーコード化する方法だ。一次元ずつバーコード添加するので、あるスポットに存在する細胞は、2種類のバーコードでラベルされることになるが、この方法により切片を処理して準備した標的核酸にバーコードを添加することが可能になった。
この方法はイエール大学で開発され、今年の2月、ヒストンコードを解析する Chip-seq を組織上で行う方法、Spatial-Cut&Tag として発表された。

この方法では、リン酸化ヒストンに対する抗体を組織上で反応させた後、抗体の存在するゲノム部位にトランスポゾンをリクルートすることで、その部位をカットしてから、X、Y 軸それぞれバーコードを流し、最後は組織全体のヒストンコードを調べる方法で、職人技を超えるほど大変そうな方法だった。
今日紹介する論文も同じグループからで、今度はもう少し単純な Atac-seq をこの方法と組みあわせられないか調べている。タイトルは「Spatial profiling of chromatin accessibility in mouse and human tissues(マウスとヒト組織上でクロマチンアクセシビリティーの空間的プロファイルを解析する)」で、8月17日 Nature にオンライン出版された。今回の論文はオープンアクセスなので、方法については、下の論文からカットアンドペーストして以下に示す。
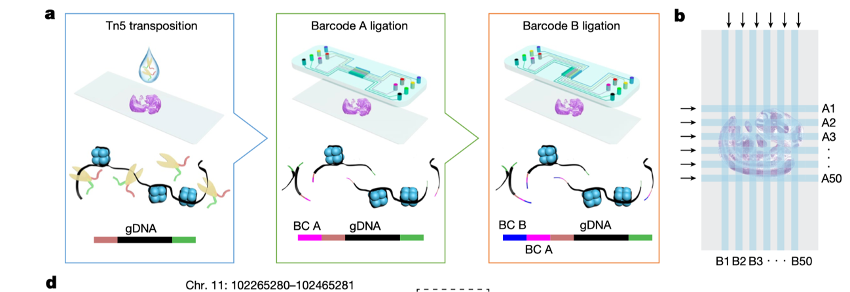
上の図からわかるように、スライドグラス上の凍結切片にトランスポゾンを反応させ、オープンクロマチンがトランスポゾンでラベルされた後切り出されるようにする。そのあと、バーコードA、バーコードBを順番に DNA に結合させ、最終的にトランスポゾンラベルとともに、A、B それぞれのバーコードから、DNA 断片の由来する場所を特定する。この DNA 断片は全て核内で生成されるので、核の組織上の位置を記録しておけば、組織とバーコードの位置を対応させることが出来る。
さて結果だが、2月の Cut&Tag の結果よりさらにわかりやすく、組織学の情報(例えば肝臓といった臓器の情報や、そこに存在する細胞の情報)が、見事にクロマチンアクセシビリティーの結果と対応し、single cell 浮遊液では得られなかった情報が満載されている。
読者の多くは single cell technology で得られる何千次元もの情報を二次元圧縮した、tSNE と呼ばれる細胞マップを覚えていると思うが、転写の様子から想像される、細胞の種類の情報が、見事に組織上で再現されテイルのを見ると、感心する。オープンアクセスなので是非自分で写真を見て欲しい(https://www.nature.com/articles/s41586-022-05094-1)。
このように将来確実に使用が拡がること間違いのない方法が完成したというのが結論だが、それだけでは素っ気ないので、2つ例を挙げてみておく。
一つはオリゴデンドロサイト系列で、成熟前は脳全体に拡がっているが、分化に伴い白質に移動すること、また人間の扁桃でT細胞がTfhへと分化する間に、エピジェネティックな変化を繰り返すことなどが見事に示されている。 現役をやめて10年になるが、この間のテクノロジーの進展は目を見張る。我が国でも、これに匹敵する様々な方法が開発されることを願っている



