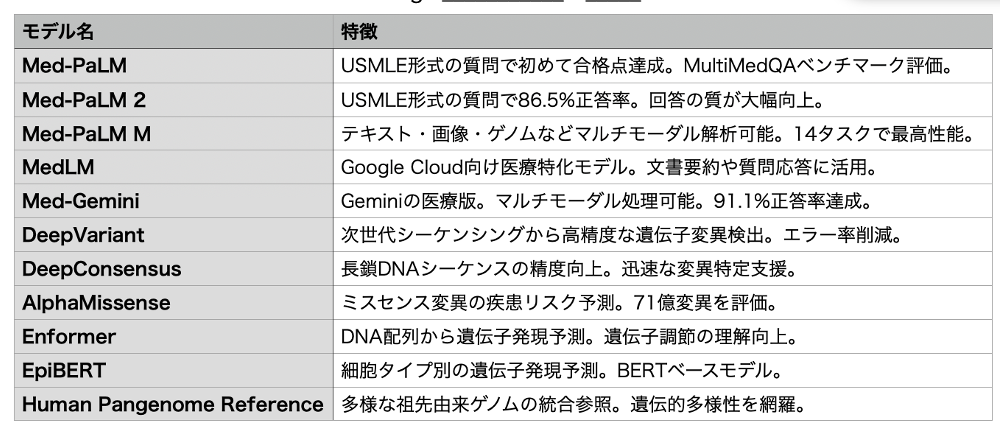
Google の2人の科学者が昨年のノーベル化学賞を受賞したことから分かるように、大規模言語モデルを進化させるためには医学生物学への進出が欠かせないと、生命科学では Alfafold2、3 だけでなくさまざまなモデルを開発している。私が目にしたモデルだけを表にしてみたが、自然言語からDNAまでまさに統合的に扱うのは医学生命科学分野だと焦点を当てているのがわかる。これにアルファフォールドや、最近紹介した鑑別診断モデルAMIEも加わり、多くがトップジャーナルへに掲載されている。
ただ Google 一人勝ちではもちろんない。それぞれのテックが生命科学に新しい可能性を求めている。例えばメタはアルファフォールドに匹敵する構造予測モデルを作成すべくスタートして、自然言語による機能記述、DNA配列、そして構造を一つの多次元空間にまとめ、言語記述から欲しいタンパク質のDNA配列を作成できる EMS3 を発表し、新しい領域を切り開き(https://aasj.jp/news/watch/26196)、なんと生命情報の研究所まで設立している。
これに対し今日紹介するのはマイクロソフトの AI 研究所からの論文で、一つのタンパク質がとりうる様々な構造を予測できるモデルの開発で、8月21日号 Science に掲載された。タイトルは「Scalable emulation of protein equilibrium ensembles with generative deep learning(タンパク質のさまざまな平衡分布を生成深層学習を用いて大規模にシミュレーションする)」だ。
アルファフォールドでタンパク質の構造予測を行うとき、最も可能性が高い一つの構造が提案されるが、実際のタンパク質はさまざまな構造変化を短時間で繰り返しており、また条件に応じて大きな構造変化をしている。これを捉えるために、スーパーコンピュータを使って自由エネルギーの計算を行いタンパク質動態を解析することが行われるが、気楽に行えるものではない。スパコンによる天気予測のように大変な計算量を伴う。
最近天気予報も普通の計算量で行えるよう大規模言語モデルが使われるが、この研究でもスパコン並の計算量が必要なシミュレーションで得られる構造変化に対応する様々な構造を予測できるモデルの開発を試みている。
これまで行われた計算シミュレーションの結果をそのままインプットしたモデルも作成できるが、データが少なすぎる。そこで、AlphaFold2 に目をつけた。以前紹介したように構造予測という点では AlphaFold2 は AlphaFold3 に道を譲った感がある。これは、AlphaFold2 がその中核の Evoformer と呼ばれる一つのタンパク質の構造を制限する進化的変化を数多くのタンパク質を比較してコンテクストとして取り出し構造予測をするためで、どうしてもタンパク質のみに構造予測が限られる。この縛りを緩めて配列自体にある構造的制約を計算することで、進化に関わらない分子との関係も自由に予測できる利点が AlphaFold3 にはある。
一方で、Evoformer は一つのアミノ酸配列を他の少しづつ違う相同分子と比べることで、どのような構造がありうるかを反復的にアップデートしながら構造を決めている。この AlphaFold2 から計算される反復的アップデートされる表象を、今度は AlphaFold3 で、進化的コンテキスト非依存的に構造化すると、一つのアミノ酸配列に対する数十種類の構造が得られる。このような Evoformer と diffusion を組み合わせた膨大なタンパク質構造データを学習させる。
これとともに、実際のタンパク質構造計算による200ms時間内の構造変化のシミュレーション結果も学習させるが、データの数は数千種類にとどまっている。一つの配列に何十種類もの異なる構造をインプットするという点では同じだが、これは物理的計算が行われた結果なので信頼でいるデータとして扱える。
最後に、たんぱく質の構造的安定性について計算した50万種類のデータをファインチューニングに使っている。(注:たんぱく質の安定性と AlphaFold2 を組み合わせる方法は、突然変異の効果を調べるGoogle Missense でも用いられている。アルゴリズムに物理法則を反映させる一つの方法のようだ)。
これら2種類のプレトレーニングと1種類のファインチューニングの結果できたモデルでは、
- RAS の GTP と GDP 結合型の構造や、CAM-KinaseII の autoinhibition による構造変化などを例に、大きく変化した構造を予測できることを示している。
- また、通常は数ヶ月もかかわる分子動態の計算シミュレーションを、小規模の GPU だけで再現できる。
- そして、たんぱく質の安定性を自由エネルギーの数値として推定できる。これは Google Missense と同じような遺伝子変異の機能解析にも使える。
以上が結果で、過去のデータを学習することで、計算に時間とコストのかかるタンパク質の動態シミュレーションを手軽に行えるようになったという話になる。
同じようにEvoformer+Diffusionを用いたタンパク質動態解析法が、中国のマイクロソフト研究所からちょうど一年前に発表されている。今回のモデルの方がスケールアップし使いやすいと思うが、異なるマイクロソフトのグループが独立によく似た方向の研究をしていることに驚いた。
このように世界を席巻しているテックは生命科学をそれぞれ独自の方法で取り込むことで、AI を進化させているのがよくわかる論文だった。現在渋谷に行くと AI センターが林立しているが、我が国でDNAを情報媒体として新しい AI モデルを目指すグループがどの程度存在しているのか気になる。少なくともトップジャーナルの論文で見る限り、テック、アカデミアを問わず日本からDNA やアミノ酸配列を媒体とした新しいモデル開発論文をまだ目にすることはない。しかし、DNAから脳、そして言語から AI と繋がってきた以上、言語以降の AI だけを扱っていては、人の後を追いかけるだけになる。我が国の若手研究者がこの分野で活躍することを願っている。



